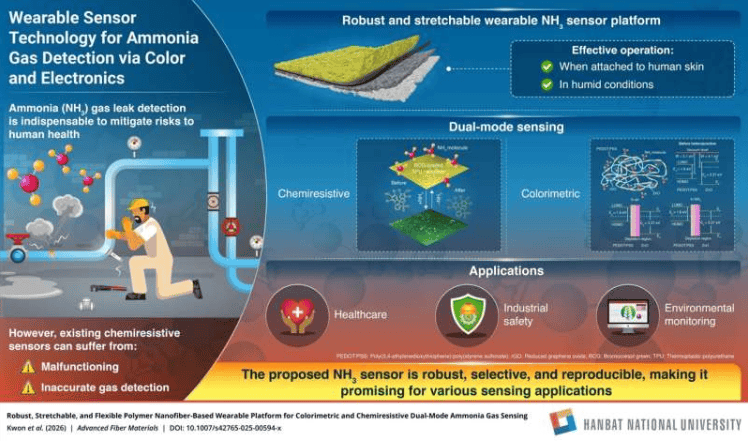
Pesquisadores japoneses desenvolveram uma nova estrutura computacional que permite inferir modelos atômicos tridimensionais de proteínas dinâmicas a partir de dados de imagens topográficas obtidas por microscopia de força atômica de alta velocidade (HS-AFM). Essa técnica, desenvolvida por uma equipe de pesquisa liderada por Holger Flechsig, do Instituto Nano de Ciências da Vida da Universidade de Kanazawa, e Florence Tama, da Universidade de Nagoya, foi publicada no periódico ACS Nano. (Imagem: diagrama de ajuste flexível do BioAFMviewer, inferindo a dinâmica biomolecular em escala atômica a partir de dados de imagens de microscopia de força atômica de alta velocidade (HS-AFM).)
A microscopia de força atômica de alta velocidade (AFM) é uma técnica experimental atualmente disponível para a observação direta da dinâmica de proteínas. No entanto, sua resolução espacial limitada dificulta o fornecimento direto de informações funcionais em nível atômico sobre biomoléculas. Para superar essa limitação, a equipe de pesquisa empregou um método de ajuste flexível computacionalmente eficiente, desenvolvido pela equipe de Tama. Esse método simula a dinâmica conformacional de estruturas proteicas estáticas conhecidas para identificar o modelo atômico que melhor corresponde às imagens experimentais de AFM.
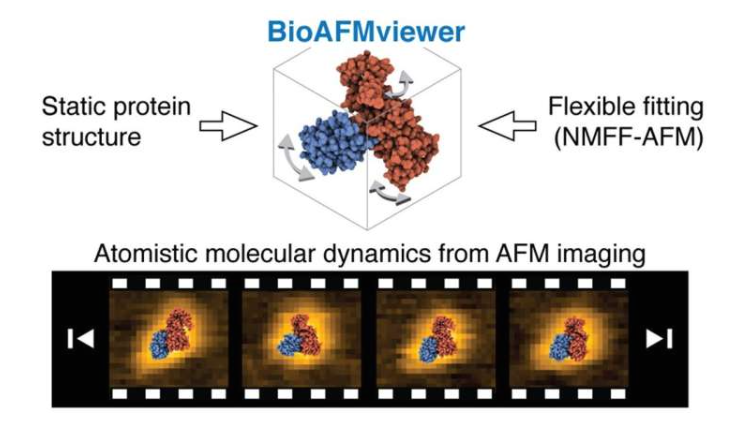
Esta estrutura computacional foi integrada à plataforma de software BioAFMviewer, mantida pela equipe de Flechsig, fornecendo um fluxo de trabalho simples para a análise de dados de imagens de AFM. Uma equipe de pesquisa analisou dados de microscopia de força atômica (AFM) de alta velocidade para uma variedade de proteínas e demonstrou que um método de ajuste flexível pode inferir modelos atômicos que incorporam movimentos de grande amplitude, melhorando significativamente a compreensão da dinâmica conformacional funcional em medições com resolução limitada.
O software BioAFMviewer apresenta alta eficiência computacional e pode ser aplicado a grandes conjuntos de proteínas. A equipe demonstrou a eficácia deste método usando um filamento de actina de 4 megadaltons composto por aproximadamente 280.000 átomos como exemplo. Com base em dados de filmes topológicos de AFM de alta velocidade, a equipe reconstruiu com sucesso filmes atômicos moleculares da dinâmica de proteínas envolvendo transições conformacionais funcionais.
Este software permite um ajuste flexível computacionalmente eficiente, integrando dados estruturais existentes e modelagem molecular com experimentos, abrindo novas possibilidades para análises em larga escala de dados de imagens de moléculas individuais. Ao aproveitar ao máximo o poder interpretativo do AFM de alta velocidade, espera-se que esta técnica promova uma compreensão mais profunda dos processos biológicos em nanoescala.
O método de AFM de ajuste flexível de padrão canônico utiliza análise iterativa de padrões canônicos computacionalmente eficiente para simular mudanças conformacionais de grande amplitude, identificando assim modelos atômicos dinâmicos que melhor representam as imagens topográficas medidas. O projeto BioAFMviewer, iniciado por Holger Flechsig em 2020, visa integrar dados de modelagem e estrutura biomolecular de alta resolução para analisar medidas de microscopia de força atômica com resolução limitada. O software fornece uma interface amigável para visualização e simulação biomolecular de imagens de microscopia de força atômica, com métodos de ajuste flexível significativamente aprimorados por meio da computação paralela.