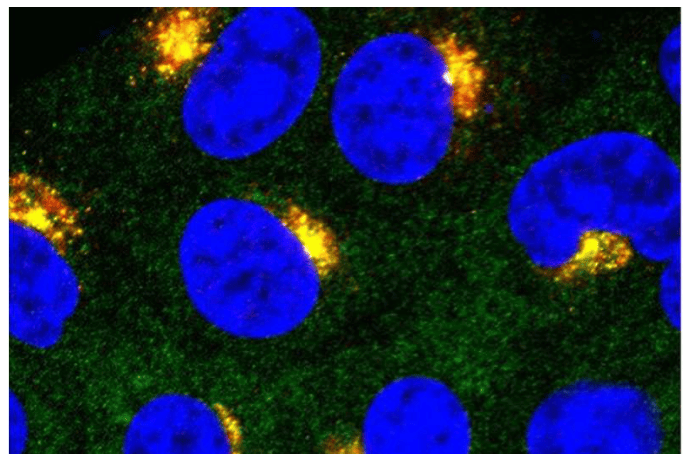
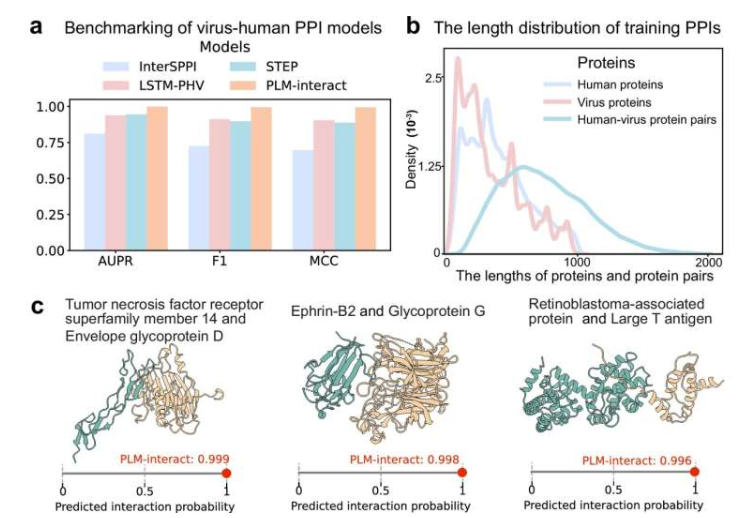
Uma equipe de pesquisa da Universidade de Glasgow desenvolveu um novo modelo de linguagem proteica, o PLM-Interact, que pode prever com eficácia as interações entre proteínas e analisar os efeitos de mutações. Este estudo, publicado na Nature Communications, utiliza recursos de supercomputação para fornecer novas ferramentas para o estudo de mecanismos de doenças e a descoberta de alvos para fármacos. Resultados de benchmark de um modelo de interação vírus-proteína humana (PPI).
Liderada pelo Dr. Ke Yuan, da Escola de Ciências do Câncer, pelo Professor Craig Macdonald, da Escola de Ciência da Computação, e pelo Professor David L. Robertson, do Centro de Virologia, a equipe de pesquisa desenvolveu o modelo de linguagem proteica PLM-Interact, baseado em um modelo de linguagem em larga escala. Treinado em mais de 421.000 pares de interações entre proteínas humanas, o modelo demonstrou desempenho preditivo superior aos modelos existentes.
O Dr. Ke Yuan afirmou: "Originalmente usado para estudar leis naturais, o supercomputador DiRAC nos ajudou a desenvolver novos modelos para explorar interações proteicas. Colegas da Escola de Ciência da Computação forneceram suporte à modelagem de linguagem, e os recursos computacionais do DiRAC nos permitiram concluir esse trabalho com mais eficiência." O treinamento deste modelo de linguagem proteica envolve mais de 650 milhões de parâmetros, e a construção do modelo foi acelerada usando o cluster de GPU na instalação de computação de alto desempenho do DiRAC no Reino Unido.
O modelo de linguagem proteica PLM-Interact (PLM-Interact) atinge uma precisão de 16% a 28% maior na previsão de interações proteicas do que outros modelos de IA líderes. O modelo previu com sucesso cinco interações proteicas relacionadas a funções biológicas importantes, enquanto outras ferramentas conseguiram prever apenas uma. Estudos também demonstraram que o PLM pode identificar com precisão os efeitos de mutações nas interações proteicas, incluindo aquelas que contribuem para doenças genéticas e câncer.
Os pesquisadores treinaram ainda mais o modelo usando dados de interação de 22.383 proteínas humanas e virais. O PLM-Interact também apresentou bom desempenho na previsão de interações entre vírus e proteínas hospedeiras, demonstrando seu potencial para aplicação em pesquisas virais. O professor David L. Robertson observou: "A pandemia de COVID-19 destacou a urgência de compreender as interações entre vírus e hospedeiros. Ferramentas como o PLM-Interact podem nos ajudar a entender melhor a emergência viral e o risco de doenças."
O desenvolvimento deste modelo de linguagem proteica fornece uma nova plataforma para a previsão precisa e em larga escala de interações proteicas, o que desempenhará um papel vital em pesquisas futuras sobre mecanismos de doenças e desenvolvimento terapêutico.