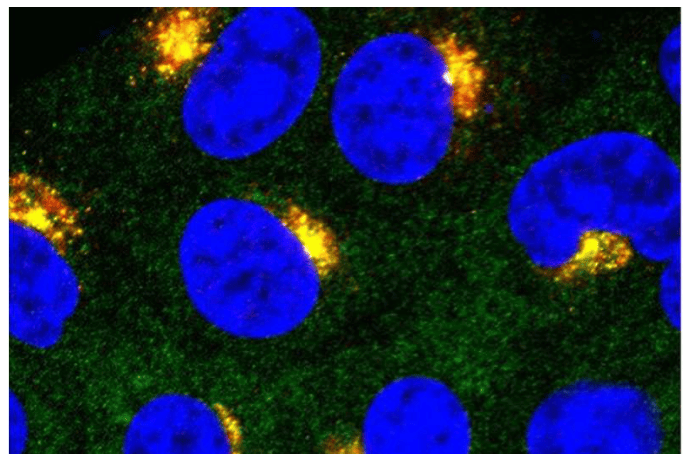
Uma equipe conjunta do Laboratório Europeu de Biologia Molecular e da Universidade de Uppsala publicou suas descobertas na *Nature Communications*, demonstrando pela primeira vez o processo dinâmico de dobramento de moléculas de RNA usando métodos integrativos de biologia estrutural. Este estudo da estrutura do RNA, empregando microscopia crioeletrônica, espalhamento de raios X em pequenos ângulos e simulação molecular, registrou o processo completo de ribozimas auto-splicing, desde a montagem do dobramento até a realização funcional. A figura mostra mapas de densidade crioeletrônica de dois estados conformacionais extremos de íntrons de classe II durante o dobramento, obtidos através do movimento dinâmico contínuo de seus motivos helicoidais estruturados.
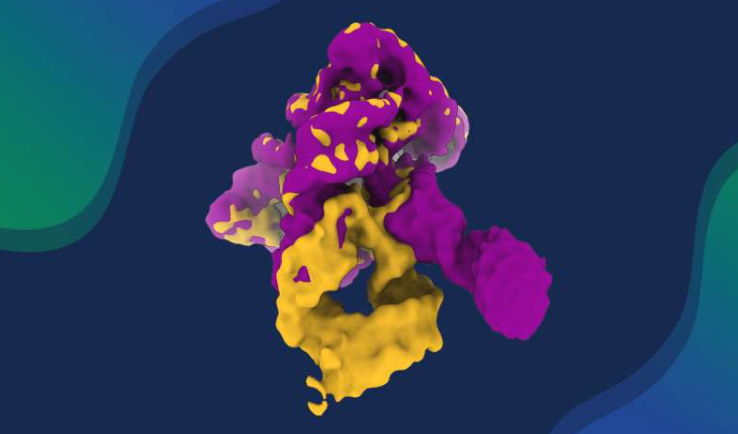
O líder da equipe, Marco Marcia, afirmou: "Este trabalho é promissor para moldar a aplicação da inteligência artificial na predição da estrutura do RNA." Ao analisar centenas de milhares de moléculas individuais de RNA, os pesquisadores reconstruíram estados conformacionais intermediários que são difíceis de capturar em estudos estruturais estáticos tradicionais, revelando o mecanismo molecular pelo qual os domínios do RNA se montam em uma sequência específica.
Dados experimentais mostram que o domínio D1 atua como um arcabouço central, regulando todo o processo de dobramento, com suas mudanças conformacionais guiando a adição sequencial de outros domínios. "Por meio de esforços contínuos e extensa triagem sob microscopia eletrônica, finalmente conseguimos observar a dinâmica complexa do RNA", observou Shekhar Jadhav, ex-pesquisador do EMBL. Esse mecanismo de montagem ordenada impede que as moléculas de RNA entrem em estados mal dobrados.
Uma solução inovadora de processamento de imagens, desenvolvida pelo Centro de Biologia de Sistemas Estruturais de Hamburgo, fornece suporte técnico para a captura de conformações transitórias do RNA. A professora Maya Topf, do centro, afirmou: "Isso demonstra de forma brilhante como a inovação computacional e dados de microscopia crioeletrônica de alta qualidade podem revelar as conformações ocultas de máquinas moleculares". Combinando simulações de dinâmica molecular, a equipe de pesquisa construiu com sucesso um mapa de energia completo do processo de dobramento do RNA.
Essa pesquisa sobre a estrutura do RNA oferece uma nova perspectiva para a compreensão da evolução da vida. O eficiente mecanismo de dobramento dos ribozimas de íntrons do tipo II, ancestrais dos spliceossomos modernos, revela o caminho evolutivo da vida primitiva mediada por RNA. Com o acúmulo contínuo de dados experimentais, esta pesquisa fornecerá um parâmetro de treinamento para inteligência artificial na predição da estrutura do RNA, impulsionando o desenvolvimento de fármacos direcionados ao RNA e nanobiotecnologia.